L’intelligence artificielle pour prédire la forme des protéines
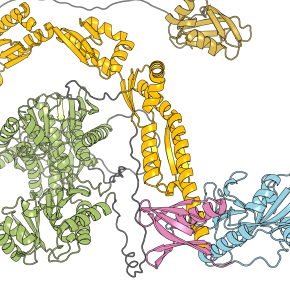
Comprendre la fonction des protéines nécessite de connaître leurs structures. Pour cela, des scientifiques ont fait appel à l’intelligence artificielle pour prédire la forme d’une classe de protéines de type « PARP » qui régulent la réparation de l'ADN, la transcription des gènes, et la réponse antivirale, mais sont aussi des cibles potentielles pour de nouvelles thérapies anticancéreuses. Cette approche, publiée dans la revue Nucleic Acids Research, pourrait s’étendre à bien d'autres familles de protéines.
Au niveau moléculaire, chaque protéine est constituée d’une chaîne d'un nombre limité de blocs, les acides aminés, qui peuvent être organisés en différentes séquences selon les gènes. Comprendre la fonction des protéines nécessite la connaissance de leurs formes tridimensionnelles (« structures »), c’est-à-dire de l’enchaînement de ces séquences.
Traditionnellement, des techniques expérimentales telles que la cristallographie des protéines sont utilisées pour déterminer ces structures. Mais, l'avènement du logiciel AlphaFold2, basé sur l'intelligence artificielle, pourrait révolutionner ce domaine en permettant de prédire, de manière fiable, la forme d'une protéine en fonction de sa séquence. Comme de nombreux autres outils d'intelligence artificielle, AlphaFold2* utilise de grands ensembles de données (dans ce cas, des ensembles de données de structures et de séquences de protéines connues) pour prédire les formes de protéines en fonction des séquences d’acides aminés qui les constituent.
Une équipe collaborative** menée par des scientifiques du Centre de biophysique moléculaire (CNRS) a ainsi testé cette approche sur la famille des protéines PARP (17 protéines chez l'homme) qui régulent la réparation de l'ADN en catalysant une modification post-traductionnelle de la protéine appelée protéine (ADP-ribosyl)ation). PARP1, la plus connue de la famille, est en effet une cible de médicaments inhibiteurs anticancéreux. D'autres PARP comme PARP5A, PARP5B et PARP14 sont récemment apparues comme des cibles potentielles pour de nouvelles thérapies anticancéreuses.
L'analyse des modèles AlphaFold2 de protéines PARP, combinée aux recherches expérimentales existantes sur la structure et la fonction de ces protéines, leur a permis de prédire et décrire la structure de chacune de ces protéines, menant à de nouvelles hypothèses sur leurs fonctionnalités dont certaines ont pu être testées expérimentalement avec succès par les auteurs. Une approche nouvelle qui pourrait maintenant s’étendre à d'autres familles de protéines.
(*) Développé par le laboratoire DeepMind de Google, AlphaFold2 est mis gratuitement à la disposition des chercheurs.
(**) Dunn School of Pathology, University of Oxford et MRC Laboratory of Molecular Biology, (Cambridge).
Rédacteur : CCdM
Référence
Updated protein domain annotation of the PARP protein family sheds new light on biological function
Marcin J. Suskiewicz, Deeksha Munnur, Øyvind Strømland, Ji-Chun Yang, Laura E. Easton, Chatrin Chatri , Kang Zhu, Domagoj Baretić, Stéphane Goffinont, Marion Schuller, Wing-Fung Wu, Jonathan M Elkins, Dragana Ahel, Sumana Sanyal, David Neuhaus & Ivan Ahel
Journal Nucleic Acids Research 2023
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad514/7199335?login=true