Arrêt sur image pour les interactions entre protéines
Contraction musculaire, transduction d’un signal, régulation de l’expression des gènes, reconnaissance des anticorps… quelques exemples parmi les innombrables processus biologiques qui reposent sur des interactions spécifiques entre protéines. Des chimistes et biologistes du CNRS ont mis au point une nouvelle méthode simple et rapide pour figer et visualiser plus efficacement ces interactions en vue de leur caractérisation. Un pas important pour la biologie structurale paru dans la revue Nature Communications.
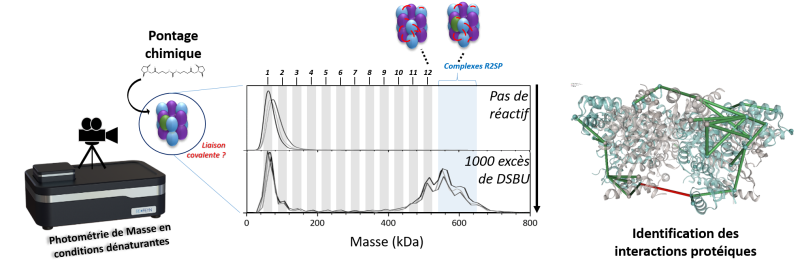
La grande majorité des processus biologiques qui régissent la vie reposent sur des interactions spécifiques entre protéines. Caractériser ces interactions constitue depuis des décennies une des pierres angulaires de la biologie structurale. Elle permet en outre de mieux comprendre certaines maladies et de jeter les bases de nouvelles approches thérapeutiques. Cette tâche s’avère cependant assez complexe car de nombreuses interactions physiologiquement pertinentes entre protéines se forment de manière transitoire. Le tour de passe-passe qui permet de « figer » ces interactions fugaces pour les soumettre à une analyse structurale consiste à les ponter chimiquement, c’est-à-dire introduire des liaisons covalentes entre les acides aminés (briques constitutives des protéines) qui interagissent. Ce pontage est fréquemment utilisé pour stabiliser des complexes formés par des protéines en vue de leur caractérisation par cryomicroscopie électronique ou spectrométrie de masse.
La première étape du protocole de pontage chimique est cruciale car son succès conditionne le résultat de la mesure biophysique qui s’en suit. En particulier, il convient d’optimiser les conditions de la réaction (température, agent de pontage, concentration…) pour garantir que les interactions figées seront préservées tout au long de l'analyse structurale ultérieure, tout en s’assurant de ne pas générer d’autres complexes biologiquement non pertinents. Pour chaque type de complexes de protéines, il convient de sélectionner le meilleur agent de pontage parmi des dizaines disponibles et de cribler les conditions de réaction chimique. Le bon fonctionnement de ce pontage, à savoir la formation des composés pontés (plus lourds) et la disparition concomitante des protéines individuelles, est ensuite vérifié par migration sur gel poreux (électrophorèse). Bien que largement disponible, facile à utiliser et peu couteuse, cette méthode de séparation est très chronophage et peu efficace car elle ne donne qu’une estimation grossière de la taille (masse) des composés formés. Des méthodes biophysiques plus précises et rapides pour identifier les espèces stabilisées de manière covalente lors du pontage chimique de protéines sont donc essentielles pour faire progresser les études de biologie structurale.
Dans ce contexte, des chimistes du l’Institut pluridisciplinaire Hubert Curien (CNRS/Université de Strasbourg) et de l’Infrastructure Nationale de Protéomique (ProFI), en collaboration avec des biologistes du laboratoire d’Ingénierie moléculaire, cellulaire et physiopathologie (CNRS/Université de Lorraine) et des scientifiques portugais, se sont tournés vers la photométrie de masse (MP), une technique émergente qui permet d’étudier des protéines ou complexes protéiques directement en solution, rapidement et sans préparation d’échantillon. Cette technique utilise le contraste résultant de l'interférence destructive entre la lumière diffusée et la lumière réfléchie par des biomolécules en solution lorsqu'elles sont irradiées par une lumière laser visible. Comme l'intensité du contraste est proportionnelle à la masse, la MP peut être utilisée pour estimer la masse de biomolécules après un étalonnage approprié avec des molécules de référence. Cette technique simple et rapide permet en outre de quantifier de façon relative les différentes espèces présentes dans une solution en fonction de leur masse, et donc, pourquoi pas, de suivre l’évolution d’une réaction de pontage entre deux protéines.
C’est ce que sont parvenus à réaliser les scientifiques qui ont développé un protocole basé sur la photométrie de masse en conditions dénaturantes qui permet l’analyse de complexes protéiques covalents et permet le criblage simple, rapide, sensible et reproductible des conditions de pontage chimique entre protéines. La résolution en masse est suffisante pour capturer non seulement le complexe final, mais aussi tous les sous-complexes transitoires stabilisés par le pontage. Ces résultats, publiés dans la revue Nature Communications, démontrent la puissance de la photométrie de masse pour accélérer les études de biologie structurale et révéler des informations auparavant inaccessibles sur les complexes protéiques étudiés.
Rédacteur: AVR
Référence
Denaturing mass photometry for rapid optimization of chemical protein-protein cross-linking reactions
Hugo Gizardin-Fredon, Paulo E. Santo, Marie-Eve Chagot, Bruno Charpentier, Tiago M. Bandeiras, Xavier Manival, Oscar Hernandez-Alba & Sarah Cianférani
Nature Communications 2024
https://doi.org/10.1038/s41467-024-47732-4