Mieux comprendre les hélicases à des fins thérapeutiques
Les hélicases, protéines essentielles dans la transcription ou la réplication de l’ADN, pourraient être exploitées à des fins thérapeutiques ou biotechnologiques. Encore faudrait-il mieux les connaître ! Pour cela, des scientifiques du Centre de biophysique moléculaire (CNRS) proposent une approche originale de criblage enzymatique qui permettra de caractériser le fonctionnement spécifique des hélicases à partir de très grandes banques de séquences d’acides nucléiques naturelles ou synthétiques. Ces résultats font l’objet d’un article dans la revue PNAS.
Les hélicases, grande famille de protéines, sont des protéines qui transforment l’énergie d’hydrolyse de l’ATP* en travail physique pour remodeler les structures d’acides nucléiques et/ou les complexes entre acides nucléiques et protéines. Malgré le rôle établi des hélicases dans le métabolisme cellulaire et dans certaines maladies (infections, cancers, etc.), les séquences d’ADN ou d’ARN à partir desquelles les hélicases peuvent agir, et la façon dont ces séquences cibles (« substrats ») sont choisies restent souvent incertaines, voire inconnues. Assez mal caractérisées, ces enzymes sont ainsi encore trop peu exploitées à des fins thérapeutiques ou biotechnologiques.
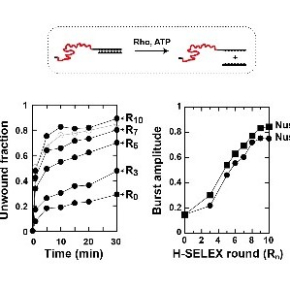
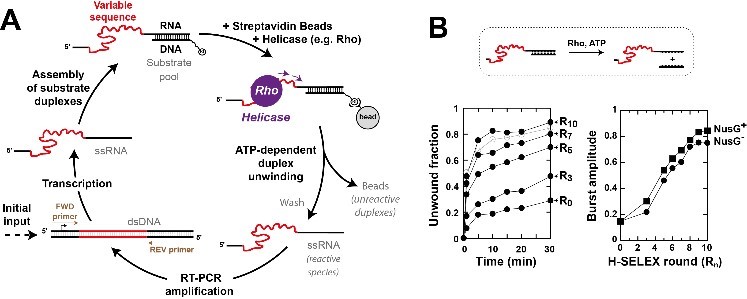
Des scientifiques du Centre de biophysique moléculaire (CNRS) à Orléans ont développé une nouvelle approche de criblage enzymatique, appelée Helicase-SELEX, pour identifier et caractériser les substrats spécifiquement ciblés par les hélicases à partir de très grandes banques de séquences d’acides nucléiques naturelles ou synthétiques. En utilisant l'hélicase Rho comme modèle, ils ont découvert ~3300 séquences de substrats pour Rho chez Escherichia coli, une bactérie qui réside dans le tube digestif de l’homme. En identifiant d’autres séquences sensibles à la présence d’un partenaire naturel (protéine NusG) ou exogène (sérotonine), les scientifiques montrent qu’Hélicase-SELEX permet la découverte de substrats inductibles, c’est à dire qui ne sont utilisés par l’hélicase qu’en présence de ce partenaire. Ainsi, Hélicase-SELEX peut être facilement adaptée à divers contextes expérimentaux et, potentiellement, à la caractérisation de nombreuses hélicases.
Ces premiers résultats, obtenus avec une hélicase modèle, montrent l’intérêt d’Helicase-SELEX pour caractériser et exploiter ensuite les propriétés des hélicases en biologie fondamentale pour l’identification des cibles physiologiques et des mécanismes moléculaires. En biologie synthétique, on pourrait envisager leur utilisation comme commutateurs génétiques** ou biocapteurs. L’automatisation du procédé de criblage sur un robot dédié -en cours au CBM- devrait permettre d’optimiser encore son exploitation.
* L’adénosine triphosphate, ou ATP, est un nucléotide qui fournit l'énergie nécessaire aux réactions chimiques du métabolisme.
** Un commutateur génétique permet de contrôler l’expression d’un gène en fonction de la présence d’une molécule ‘inductrice’. Les auteurs ont par exemple montré que des substrats de l’hélicase Rho inductibles à la sérotonine pouvaient remplir ce rôle en régulant la transcription d’un gène de façon Rho-dépendante.
Rédacteur : CCdM
Référence
Delaleau M., Eveno E., Simon I., Schwartz A & Boudvillain M.
A scalable framework for the discovery of functional helicase substrates and helicase-driven regulatory switches
PNAS 2022