La structure de la face cachée de l’enzyme NADPH oxydase dévoilée
L’activation de la NADPH oxydase, enzyme majeure du système immunitaire, dépend de l’assemblage des sous-unités qui la constituent, soit trois unités cytosoliques et deux unités membranaires. Grâce à une approche originale basée sur des techniques d’imagerie en cellule vivante et la modélisation 3D, une équipe du Laboratoire de chimie physique d’Orsay (CNRS/Université Paris-Sud/Université Paris-Saclay) et leurs collaborateurs proposent le premier modèle structural complet de la partie cytosolique de l’enzyme. Ces travaux qui marquent le point de départ pour élucider les étapes d’activation de cette enzyme cruciale sont publiés dans The Journal of Biological Chemistry.
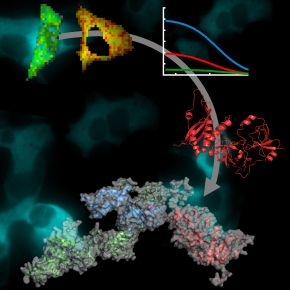
L’enzyme NADPH oxydase permet aux cellules phagocytaires du système immunitaire de produire des substances toxiques pour détruire les pathogènes (virus, bactéries, etc.) dans le compartiment où elles les séquestrent, après les avoir capturés. Si cette enzyme est essentielle aux mécanismes de défense de l’organisme, sa dérégulation a par ailleurs de lourdes conséquences : son hyperactivité est un facteur aggravant, voire déclenchant, de nombreuses maladies inflammatoires telles que les bronchites chroniques. Afin de mieux comprendre les mécanismes qui sous-tendent l’activation de l’enzyme, les chercheurs du Laboratoire de chimie physique (LCP) d’Orsay se sont penchés sur l’organisation structurale de trois des cinq sous-unités qui la composent, à savoir ses trois unités cytosoliques.
Lorsque le phagocyte leur en donne l’ordre, les sous-unités de la NADPH oxydase se rassemblent pour former l’enzyme active. Son activité résulte donc des interactions entre ses trois sous-unités cytosoliques déjà assemblées au repos et les deux sous-unités membranaires. Les techniques utilisées jusqu’à présent n’ont pas permis d’analyser complètement la structure des unités cytosoliques, leur grande taille et leur flexibilité les rendant difficiles à étudier in vitro. Les chercheurs du LCP ont contourné cet obstacle en développant une stratégie d’analyse directement en cellules vivantes. En étiquetant les sous-unités cytosoliques avec des protéines fluorescentes et en utilisant la complémentarité de deux techniques de microscopie (FRET-FLIM et FCCS), ils ont défini comment ces unités interagissent entre elles et évalué la distance qui les sépare. En complétant cette approche avec les données partielles obtenues in vitro, les chercheurs ont mis au point un modèle topologique complet des trois sous-unités cytosoliques, qui décrit leur position et leur orientation les unes par rapport aux autres.
Non seulement ce résultat constitue la première étape pour accéder aux interactions successives qui régissent l’activation de l’enzyme, mais il permet aussi d’envisager des pistes thérapeutiques pour pallier son hyperactivité pathologique. Celles-ci consisteraient à inhiber les interactions entre sous-unités pour bloquer l’assemblage nécessaire à l’activation de l’enzyme. Par ailleurs, la stratégie mise au point peut désormais être transposée pour étudier les interactions d’autres protéines contenant des domaines flexibles.
Référence
Quantitative live-cell imaging and 3D modeling reveal critical functional features in the cytosolic complex of phagocyte NADPH oxidase. Cornelia S Ziegler, Leila Bouchab, Marc Tramier, Dominique Durand, Franck Fieschi, Sophie Dupré-Crochet, Fabienne Mérola, Oliver Nüße and Marie Erard.
J. Biol. Chem. 10 janvier 2019 294: 3824